Патогенная бактерия использует простой аминокислотный код для управления генами своих жертв
|
Бактерия Xanthomonas вводит в растительные клетки белки TAL, которые проникают в ядро и регулируют активность генов. Германские и американские исследователи независимо друг от друга расшифровали аминокислотный код, при помощи которого белки TAL безошибочно распознают регуляторные участки нужных им генов. Открытие имеет важное значение для биотехнологии, поскольку позволяет целенаправленно проектировать белки, присоединяющиеся к строго определенным участкам ДНК.
Строение белковых молекул «записано» в ДНК при помощи стандартного генетического кода, расшифрованного в начале 1960-х годов. То, в каких обстоятельствах и с какой интенсивностью должны работать те или иные гены, «записано» в ДНК другими, гораздо более сложными кодами. Эта информация, расшифрованная пока лишь частично, управляет взаимодействием двойной спирали ДНК с разнообразными регуляторными белками (см. факторы транскрипции).
Общий принцип действия белков — регуляторов транскрипции состоит в том, что они находят на ДНК определенную последовательность нуклеотидов (свой «сайт связывания»), прикрепляются к нему и тем самым либо увеличивают, либо снижают активность близлежащего гена. Понятно, что в аминокислотной последовательности белка-регулятора каким-то образом «закодирована» нуклеотидная последовательность сайта связывания, иначе регуляторы просто не смогли бы найти и опознать свой сайт. Однако расшифровать этот код очень сложно. Ничего похожего на линейное соответствие между последовательностью аминокислот в молекуле белка-регулятора и нуклеотидов в молекуле ДНК обычно не наблюдается. Это естественно, поскольку белковые молекулы функционируют не в виде одномерных нитей — цепочек аминокислот, а в виде сложных трехмерных «клубков», в которые эти нити сворачиваются (либо самостоятельно, либо при помощи специальных белков — шаперонов). Зная аминокислотную последовательность белка, в принципе можно рассчитать, в какую трехмерную структуру он свернется самопроизвольно, если ничто ему не помешает. Но эта задача технически очень сложна. Кроме того, в процесс сворачивания белковой молекулы всегда может вмешаться какой-нибудь «сторонний» фактор (например, белок может быть химически модифицирован другими белками), и тогда все результаты расчетов можно смело выбрасывать на помойку. Более того, даже зная трехмерную структуру белка, очень трудно определить, какие именно аспекты этой структуры отвечают за распознавание тех или иных нуклеотидов сайта связывания.
Древнеегипетская письменность, как известно, была расшифрована при помощи Розеттского камня, на котором один и тот же текст был написан на двух языках — египетском и греческом. У молекулярных биологов тоже есть свой «Розеттский камень» — длинный список нуклеотидных последовательностей сайтов связывания и аминокислотных последовательностей белков, распознающих эти сайты. Но задачка оказалась посложнее, чем с египетскими иероглифами, и расшифровка движется медленно. Помимо вышеупомянутых трудностей, проблема еще и в том, что разные группы регуляторных белков явно используют разные системы кодирования.
В последнем номере журнала Science сообщается об успешной расшифровке одной из этих систем. Открытие сделано независимо учеными из Университета штата Айова (США) и Галле-Виттенбергского университета имени Мартина Лютера (Германия). Оба коллектива изучают регуляторные белки TAL (transcription activator–like), при помощи которых патогенная бактерия Xanthomonas манипулирует работой генома растительных клеток. Расшифровка «регуляторного кода» в данном случае оказалась возможной благодаря тому, что эти белки, как выяснилось, используют довольно простую линейную систему кодирования.
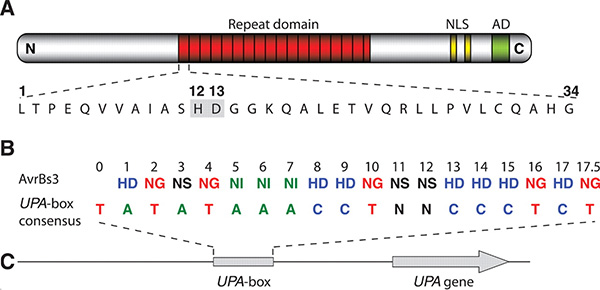
В аминокислотной последовательности белков TAL есть участок, состоящий из многократно повторяющихся 34-аминокислотных «слов». Все «буквы» (то есть аминокислоты) в этих словах одинаковы, кроме двух — 12-й и 13-й. Оказалось, что именно эти вариабельные пары букв и кодируют последовательность нуклеотидов в сайте связывания.
|
Расшифрованный код допускает неоднозначность (некоторые пары аминокислот кодируют более одного нуклеотида) и является «вырожденным» (некоторые нуклеотиды кодируются более чем одной парой аминокислот). Тем не менее этот код позволяет, зная структуру белка TAL, «вычислить» его сайт связывания, и наоборот — имея последовательность нуклеотидов, спроектировать белок TAL, который будет избирательно прикрепляться именно к такой последовательности.
| Пара аминокислот | Кодируемые нуклеотиды | |
| HD | гистидин — аспарагиновая кислота | C |
| NG | аспарагин — глицин | T |
| NI | аспарагин — изолейцин | A |
| NS | аспарагин — серин | A, C, G, T |
| NN | аспарагин — аспарагин | A, G |
| IG | изолейцин — глицин | T |
Последнее обстоятельство особенно важно с практической точки зрения. Данное открытие открывает простой и дешевый путь к искусственному проектированию и изготовлению белков, которые будут избирательно связываться с любыми интересующими нас нуклеотидными последовательностями. Такие технологии очень важны не только для научных исследований, но и для медицины — в частности, для борьбы с вирусными заболеваниями. На их основе, в принципе, можно разработать методы изъятия из генома тех или иных нежелательных фрагментов ДНК, в том числе встроившиеся вирусные геномы. В заметке Искусственный белок поможет победить ВИЧ («Элементы», 05.07.2007) рассказывалось о том, как подобную задачу долго и мучительно решали методом искусственной эволюции. Новое открытие позволит перейти от медленных «дарвиновских» технологий к более быстрому и дешевому «разумному дизайну».
Американские исследователи в своей работе ограничились биоинформационными подходами, то есть расшифровали код путем изощренного компьютерного анализа нуклеотидных и аминокислотных последовательностей (см.: М. С. Гельфанд. Что может биоинформатика). Немцы продвинулись чуть дальше и подтвердили правильность расшифровки кода экспериментально. В частности, они спроектировали и изготовили несколько новых белков-регуляторов, соединили их с молекулами зеленого флуоресцирующего белка и экспериментально показали, что они избирательно связываются только с теми последовательностями нуклеотидов, которые были «зашифрованы» в их аминокислотной последовательности при помощи новооткрытого кода.
Источники:
1) J. Boch et al. Breaking the Code of DNA Binding Specificity of TAL-Type III Effectors // Science. 2009. V. 326. P. 1509–1512.
2) M. J. Moscou, A. J. Bogdanove. A Simple Cipher Governs DNA Recognition by TAL Effectors // Science. 2009. V. 326. P. 1501.
Александр Марков